Amber
本示例说明如何在云超算平台上运行作业,平台目前支持两百多种软件,您可根据自己的需求选择自己所需的软件和计算文件;本次使用软件是以Amber为例。Amber是著名的分子动力学软件,用于蛋白质、核酸、糖等生物大分子的计算模拟。Amber也指一种经验力场(empirical force fields)。
本页目录
作业配置
| 输入文件 | initial.in |
|---|---|
| 软件版本 | Amber1.9 |
| 命令 | sbatch -N 1 -n 1 -p g-t4-1 demo-amber.sh |
| 建议硬件 | 白鲸/1卡4核15G/1总卡数 |
作业提交方式
本软件的作业提交方式有三种,分别为界面提交,命令行提交和Windows工作站提交,三种方式都可以帮你完整的提交运行您的作业。您可以点击下方的tab栏进行切换查看各种提交方式。
 命令行提交
命令行提交 Windows工作站提交
Windows工作站提交Step 1. 选择软件
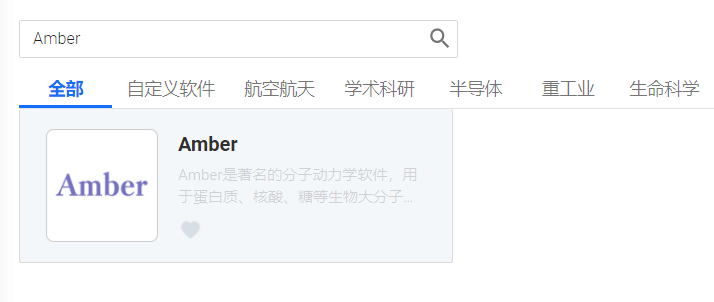
Step 2. 选择Amber模板,Amber模板有CPU并行,GPU单节点,MPI并行等。
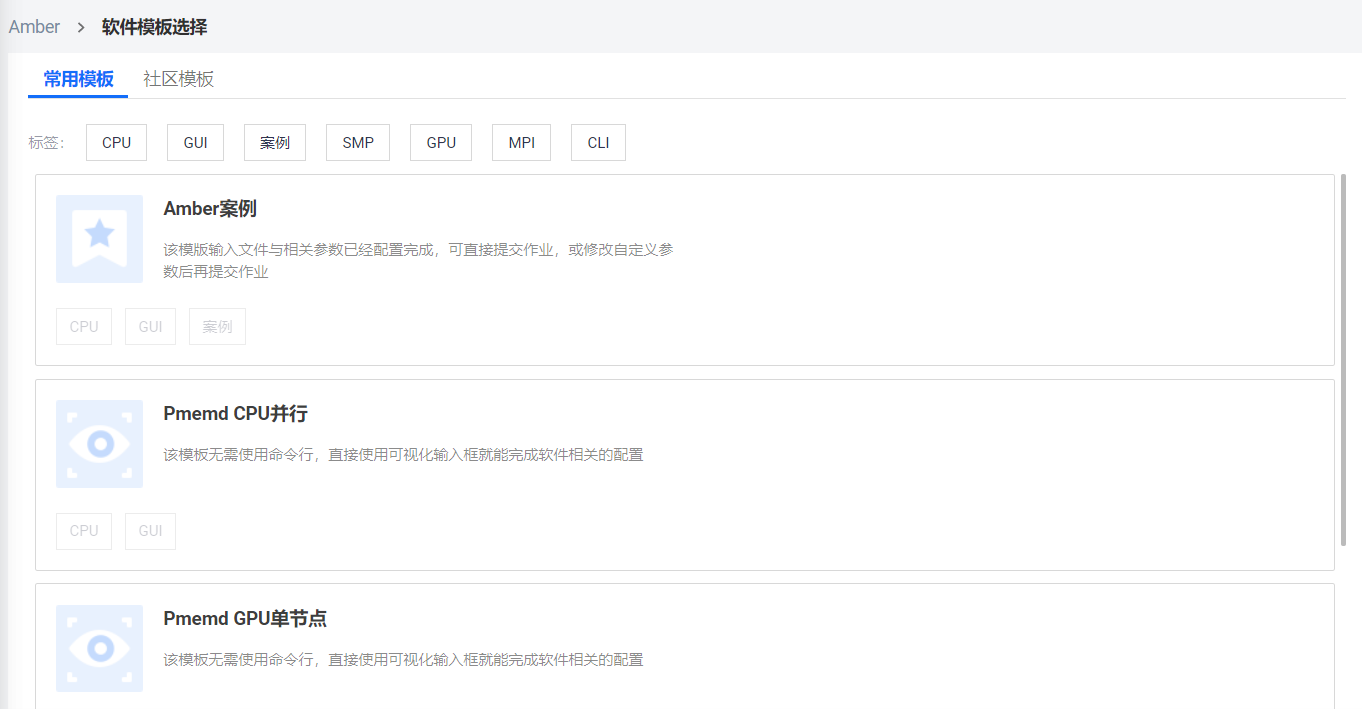
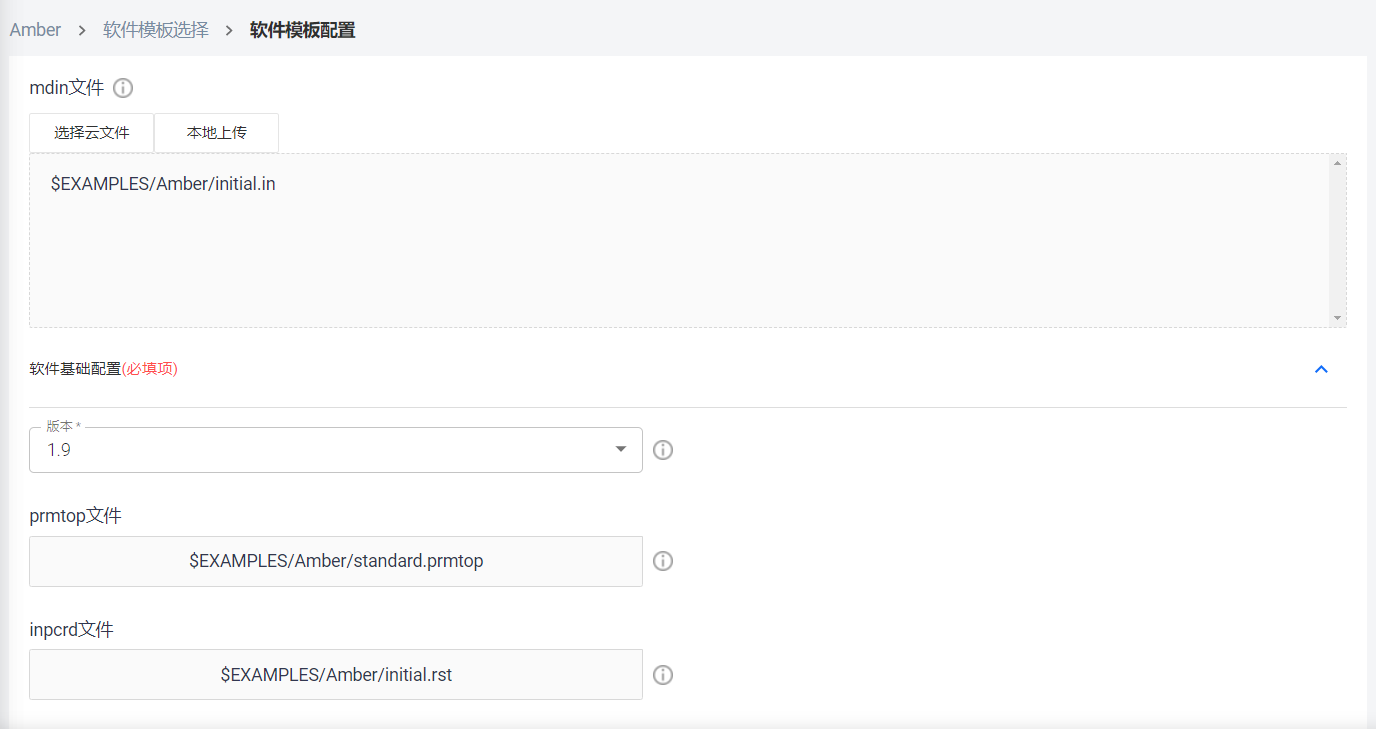
Step 3. 配置模板参数,传入输入文件。
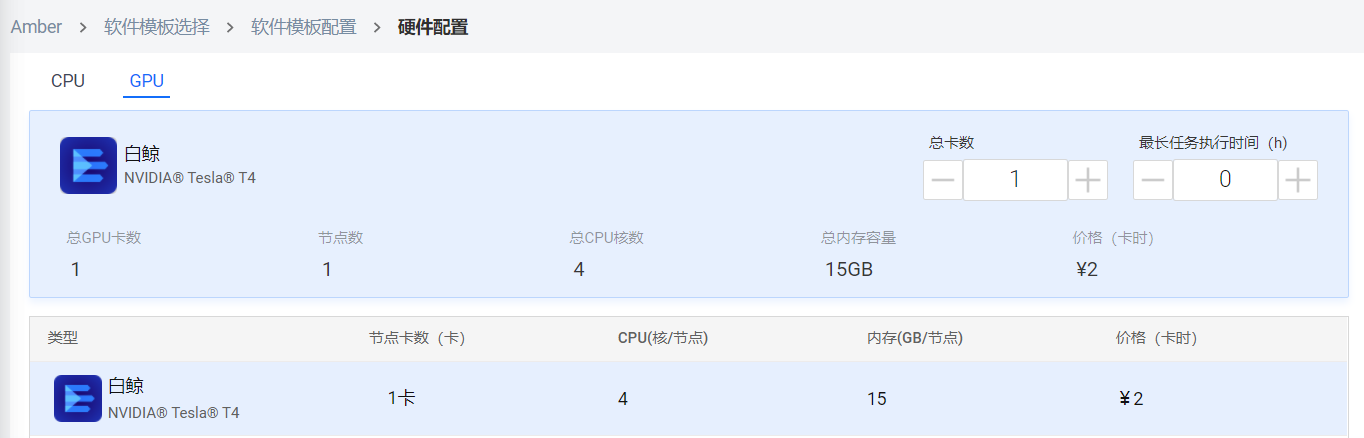
Step 4. 硬件选择白鲸,总卡数为1。
Step 5. 提交作业。
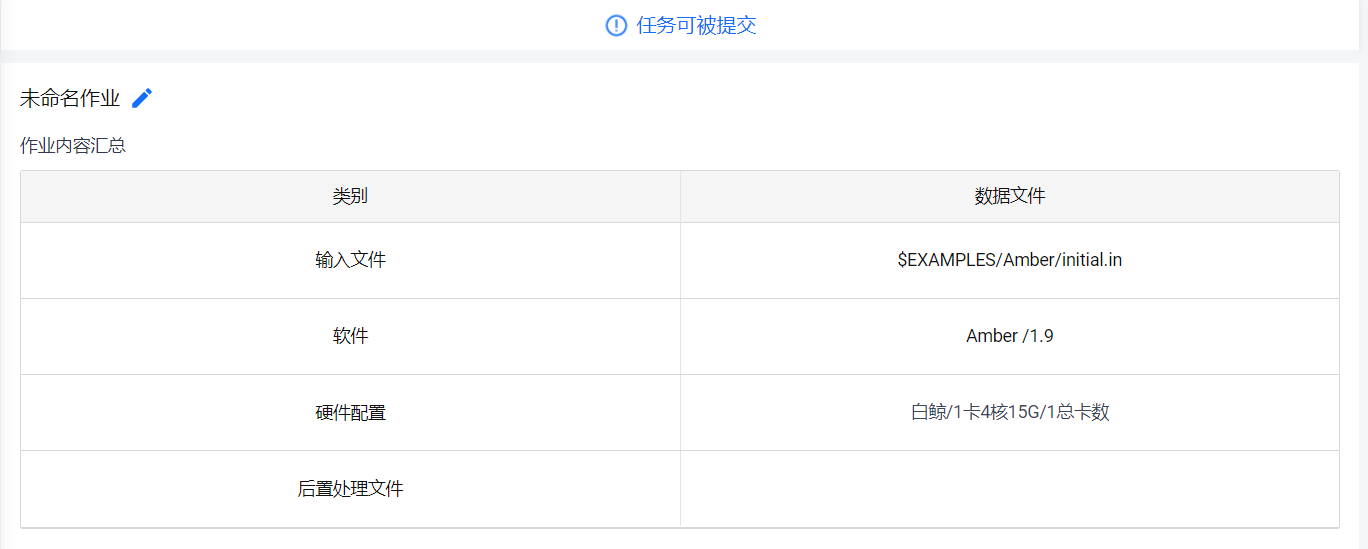
Step 6. 作业结果。可以在下方点击查看结果文件,日志文件等。
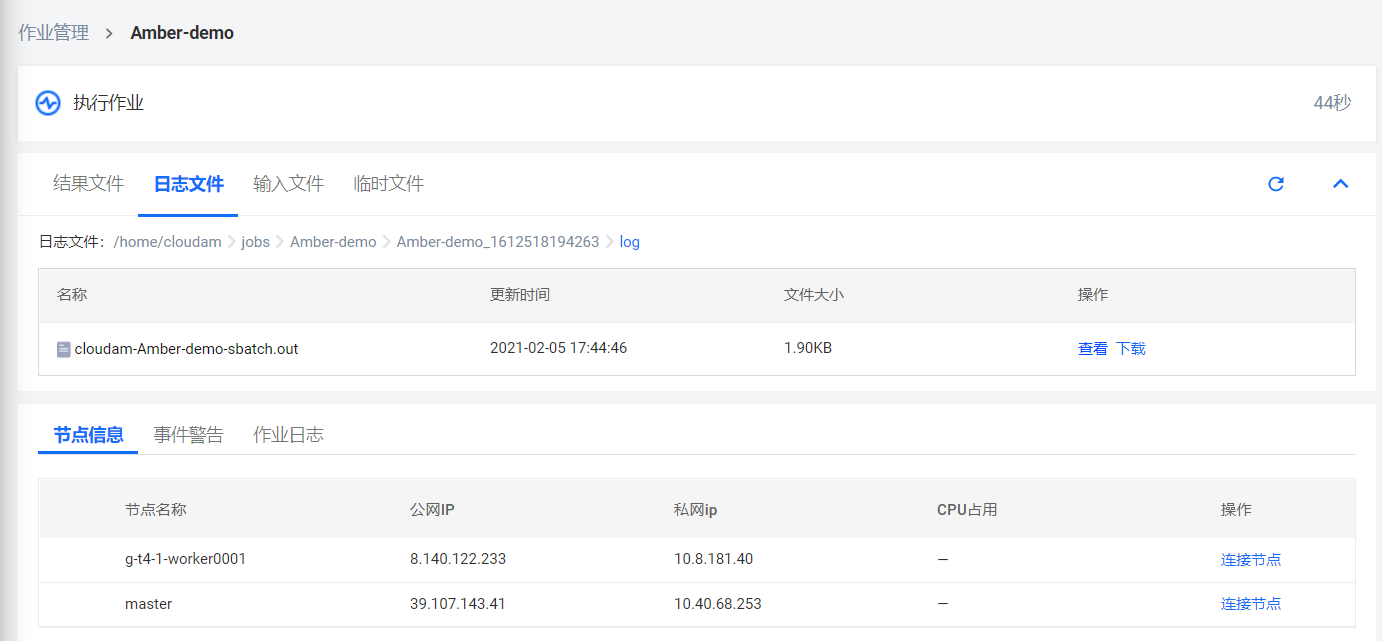
通过命令行提交作业,需要创建管理节点,创建及连接操作见作业管理节点
节点创建成功后,我们就可以在管理节点提交作业。
进入管理节点后,我们需要找到所需要的软件。具体操作见作业管理节点
之后,我们需要将之前的加载软件命令与软件运行命令结合起,创建一个名为demo-amber的shell脚本来提交作业。内容如下:
脚本创建后,我们需要输入命令启动作业,命令如下
其中,-N为节点的数量,这里输入的是1。-n为任务进程数量,这里为1。-p为选择的PARTITION,这里使用的是白鲸(g-t4-1)。
如上图所示。任务提交成功,jobid为18.
任务提交后,可以查看任务的详情,节点状态等。具体操作见作业管理节点
通过桌面工作站提交,需要创建桌面工作站节点,创建及连接操作见桌面工作站
节点创建成功后,我们就点击连接就可以来到桌面工作站页面。
进入桌面工作站节点后,通过网络,安装执行任务所需要的软件。
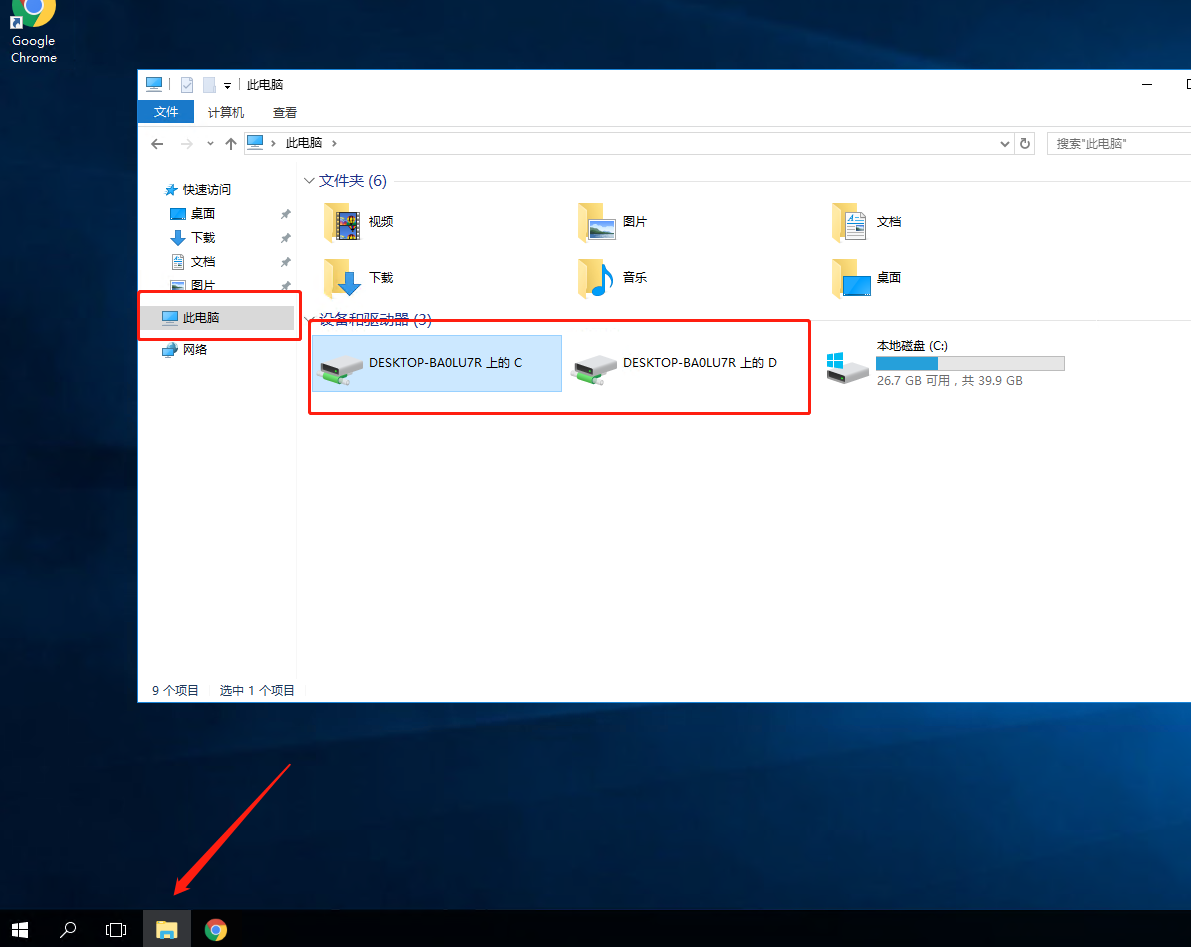
我们也可以通过本地电脑向桌面工作站传输文件。点击下方的文件栏,再点击我的电脑。可以看见多个硬盘。本地磁盘c:是当前桌面工作站的硬盘,前面的2个硬盘分布对应了我们本地电脑的c盘和d盘。这样,我们就可以通过简单的移动或者复制,把需要的文件传输到桌面工作站中。
查看作业输出文件
待作业执行完成后,我们可以在多个页面获取到作业的输出文件,详情见查看作业输出文件
