


ISV( Independent Software Vendors,以下简称ISV)计划是由北鲲云发起,秉承完全开放的生态策略,致力于与合作伙伴一起,为有高算力计算需求的用户提供更多样且更具价值的极致应用,给北鲲云用户带来更多选择及便利,也为合作伙伴提供平台推广服务及专业技术支持。我们希望与国内外计算机软件开发者成为合作伙伴,将更多的应用和服务集成到北鲲云超算平台。
港中文深圳理工学院-未来智联网络研究院-上海智峪生科联合团队(包括但不限于)带着 PointSite(蛋白质上基于深度点云模型的小分子结合区域预测软件 )入驻北鲲云超算平台,服务于生命信息行业用户,有助于提高基于结构的药物发现精度和未来潜在的靶点寻找的效率。
关于Pointsite
PointSite是一种无需模板的配体结合位点预测方法,用于精确识别蛋白质配体结合原子,它以蛋白质为中心的方式在原子水平上进行蛋白质LBS识别。具体来说,PointSite主要由三个模块组成,原子点云转换(APCT)模块、配体结合原子预测(LAP)模块以及LBS识别(LBSI)模块。
在APCT模块中,首先将原始的蛋白质结构从PDB格式转换到原子级的点云。LAP模块将原子的点云坐标转换为稀疏体积表示,而原子的特征保持不变,利用基于子流形稀疏卷积(SSC)的U-net对配体原子进行预测。由于稀疏卷积的操作,我们可以用更高的分辨率表示点云,并保持计算效率。接着,LBSI模块将点云分割的结果和以口袋为中心的方法进行结合:滤除较低概率的原子,并对剩余原子进行重新排序,从而得到更加精确的结合位点原子预测。

(PointSite工作示意图)
作为药物设计的第一环节,PointSite目前正在被运用于学术研究和药物开发中。研究结果表明,PointSite中分割出的结合位点原子可以作为一个后处理工具,通过过滤和重新排序策略来指导任何以口袋为中心的方法,以优先确定识别出的伪表面点(PSPs)。由于以口袋为中心的方法可能会输出许多假阳性结果,因此后续的优先级划分步骤可以大大提高此类工具的性能(识别成功率)。同时,在常用的DCA准则和所有口袋中心方法的原子级IoU方面可以大大提高识别准确性。因此,PointSite是基于结构的药物发现的高效工具。
实验结果
1、PointSite 识别结合位点原子的准确性
本部分的实验使用atom-IoU指标,即预测原子集合和真实原子集合的交并比来衡量各方法预测的结合位点原子的准确性。
表1列出了PointSite与其他方法在B277、DT198、ASTEX85、CHEN251、COACH420 和 HOLO4k数据集上的atom-IoU结果。从表1中可以看出,在几乎所有数据集上,PointSite方法明显优于其他方法,仅次于PointSite的方法是P2Rank。PointSite在 DT198、B277、ASTEX85、COACH420 和 HOLO4k 上的atom-IoU分数分别比 P2Rank 高 7%、11%、14%、14% 和 15%。在CHEN251数据集,也就是P2Rank的训练集上,PointSite与P2Rank也有相当的效果。
表1: 各方法在B277、DT198、ASTEX85、CHEN251、COACH420 和 HOLO4K 数据集上的识别性能比较。(以 atom-IoU (%) 表示)

2、PointSite 预测结合位点中心位置的性能
DCA指标用于描述预测结合位点(口袋)的中心与配体的任何原子之间的最小距离,本部分的实验用DCA指标来测量各方法预测结合位点中心位置的性能。
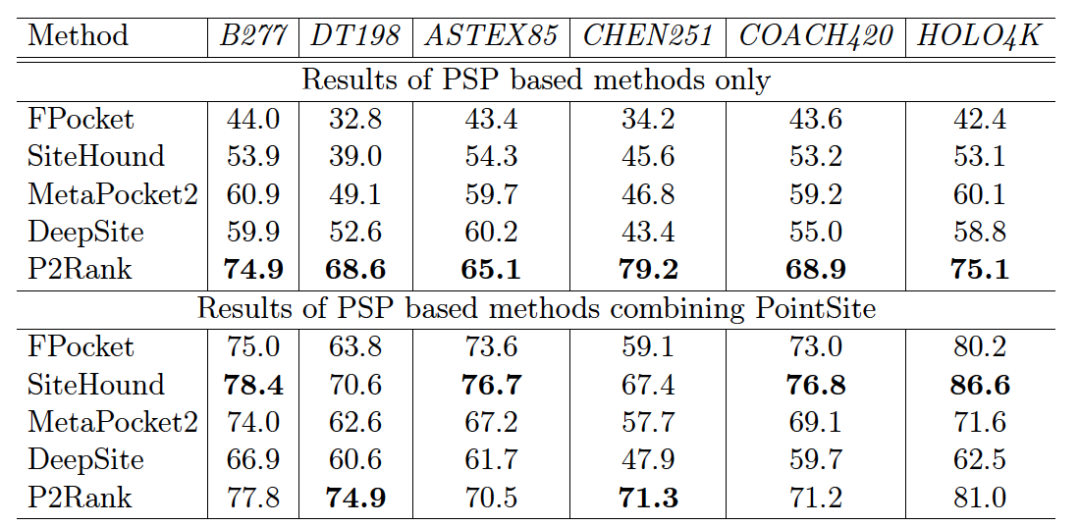
PointSite不仅可以单独使用来预测结合位点相关的原子,也可以作为一个灵活的过滤工具和其他以口袋为中心的方法结合,从而提高其他方法的预测性能。如表2所示,结合PointSite可以在几乎所有数据集上显著提高以口袋为中心的方法的DCA 分数(除了 CHEN251,是 P2Rank 的训练集)。值得注意的是,对于SiteHound方法,它的原始结果比 P2Rank 低约10-20% ,但是,在SiteHound 与PointSite结合后,它结果分别比 P2Rank 在 B277、DT198、ASTEX85、COACH420 和 HOLO4k 上的原始结果高 4%、2%、11%、8% 和 11%。在一些数据集中,如B277、ASTEX85、COACH420、HOLO4k,SiteHound的结合后结果甚至比P2Rank结合后结果还要高1%、6%、5%、6%。
表2:以口袋为中心的方法以及这些方法与PointSite结合的结果的DCA指标(阈值为4 Å)在B277、DT198、ASTEX85、CHEN251、COACH420 和 HOLO4K 数据集上的识别性能比较。
3、CAMEO数据集上的atom-IoU和DCA性能结果
为了测试我们的点云分割方法是成功地学习到了三维蛋白质结构和结合原子之间的复杂关系,还是只是简单地“记住”了训练数据。我们从CAMEO数据集中选择了难度较高的蛋白质结构,并且这些蛋白质不仅序列相似度很低 (< 25%),而且与我们的训练数据集的结构相似性度也较低(TMscore < 0.648),从而来模拟真实的药物发现场景。
如表3所示,在CAMEO数据集中,单独使用PointSite达到了 0.43 atom-IoU,分别比单独使用FPocket、Site-Hound、MetaPocket2、DeepSite 和 P2Rank提高了 18%、23%、19%、21% 和 6%,这表明PointSite具有较好的泛化性。
当以口袋为中心的方法与PointSite结合时,它们的识别精度在 atom-IoU 和 DCA 方面都有显著提高。比较典型的是FPocket 和 SiteHound,它们分别是基于几何和能量方法,而不是基于机器学习的方法(例如 DeepStie 和 P2Rank)。这些结果进一步证明了我们的以蛋白质为中心的方法与以口袋为中心的方法提取到的蛋白质的信息是相互补充的。
表3:各方法在CAMEO数据集上的atom-IoU和DCA(阈值为4 Å)指标的比较

4、案例分析
案例1:未结合-结合蛋白对(unbound-bound protein pairs)的案例
在药物设计中,预测未结合状态下的蛋白质潜在结合位点是一个重要的任务。如图3所示,左边三列是未结合-结合蛋白对1q1c-4lay(人体FKBP52的前1-260个残基的蛋白质结构)。与配体结合后,蛋白质和配体结合的区域相比于未结合时有明显的形变(见中间真实值列中的红色点云标签)。即使对于未结合的蛋白质 1q1c,我们的 PointSite 也可以预测出近似的结合位点。图3右边三列是未结合-结合蛋白对1p9r-1p9w(霍乱弧菌推定的 NTPase EpsE 的蛋白质结构),即使在对未结合蛋白结构的预测中存在更多的假阳性原子,PointSite 始终可以为蛋白结构获得近似的配体结合位点。
图2:两组未结合-结合蛋白对的预测结果的可视化效果图

案例2:SARS-CoV-2 (COVID-2019) (PDB ID: 7BTF) 的 RNA 依赖性 RNA 聚合酶 (RdRp) 和 SARS-Cov2 (PDB ID: 7BQY) 的主要蛋白酶 (Mpro)的预测效果
如图3所示,中间的一列图片显示了真实的结合位点位置,最右边的一列显示了PointSite的预测结果。PointSite不但能够预测出准确的结合位点的位置,而且其预测出的结合位点的位置也包括了核心的残基所在的位置。这些结果表明,PointSite 可以成为未来基于结构的药物设计的一个非常有潜力的工具。
图3 PointSite对SARS-CoV-2 (COVID-2019) (PDB ID: 7BTF) 的 RNA 依赖性 RNA 聚合酶 (RdRp) 和 SARS-Cov2 (PDB ID: 7BQY) 的主要蛋白酶 (Mpro)的预测结果

PointSite在北鲲云上的使用
PointSite的入驻是北鲲云ISV计划重要的第一步,也是一个良好的开端。PointSite的入驻,为北鲲云的用户提供了更多样的选择,能够帮助用户得到更精准的计算结果,提高计算效率。同时,PointSite结合北鲲云平台产品与服务卓越的安全性及灵活性,能够为用户提供更优质的服务,帮助其进一步提升整体运营能力与盈利水平。
什么是北鲲云ISV计划?
加入ISV计划,北鲲云为您提供

我们期待未来有更多的合作伙伴加入北鲲云ISV计划。